1. 访问与安装,可视化文件或SMILES代码
1.1. 访问与安装
使用Qbics-MolStar有两种方式:
直接在线访问链接:https://molstar.szbl.ac.cn/viewer
安装安装包:
点击链接进入Qbics-MolStar网站界面:https://molstar.szbl.ac.cn/download/
点击如图所示位置执行下载:
1.2. 分子可视化:本地文件
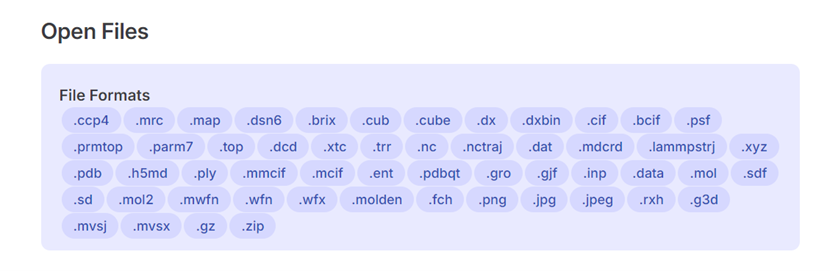
当前支持文件格式包括:.top,.cif,.gjf,.inp,.mol,.xyz,.pdb,.mwfn,.mol2等等,具体介绍详见https://molstar.szbl.ac.cn/docs/use/。
用户可以通过两种方式实现已知分子坐标的可视化(以.pdb为例):
直接拖拽文件至Qbics-MolStar界面,即可实现可视化:
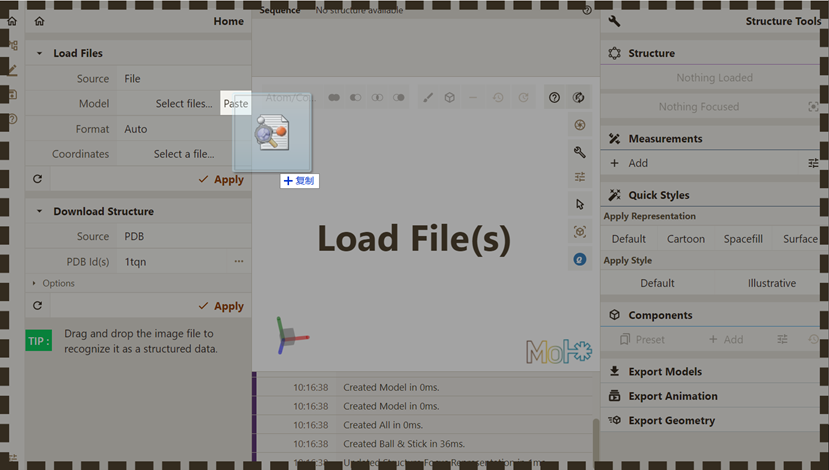
选择保存在本地的文件:
点击下方红框按钮,选择希望可视化的文件。
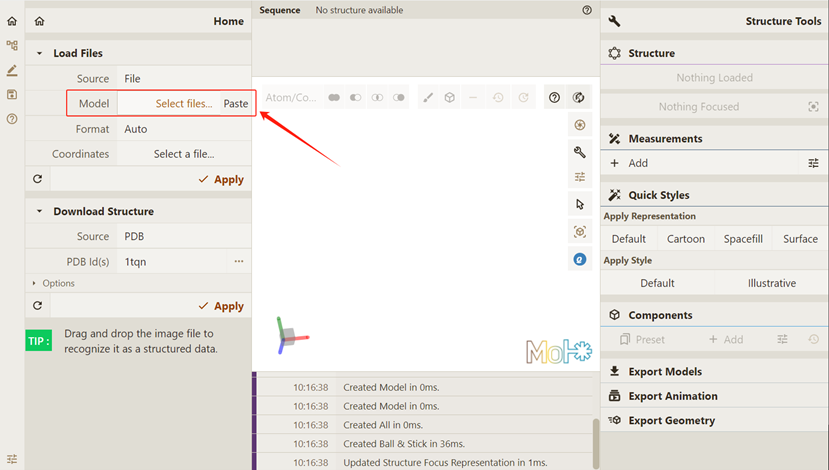
双击选中的文件(此处为c60.pdb)使其加载至Qbics-MolStar:
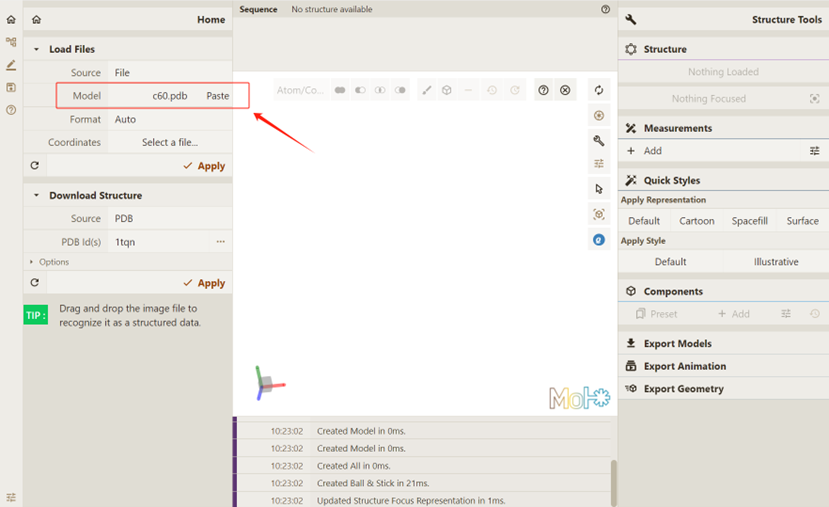
点击 Apply,使Qbics-MolStar开始渲染体系,实现可视化:
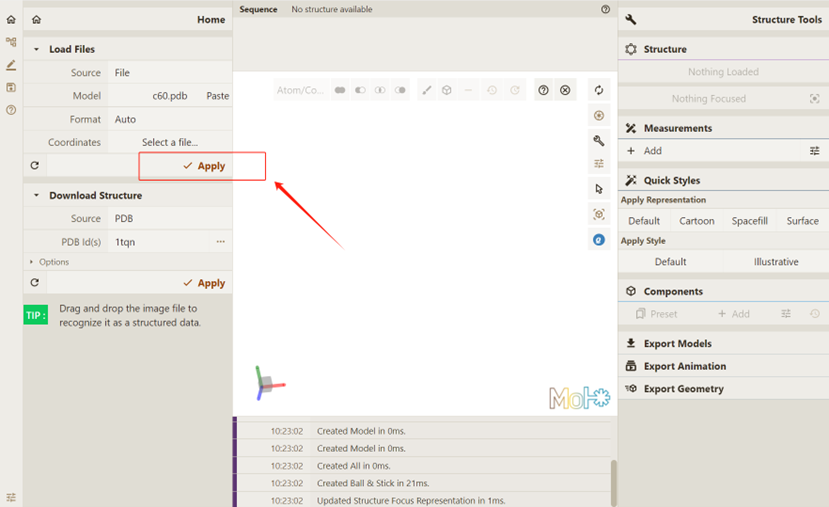
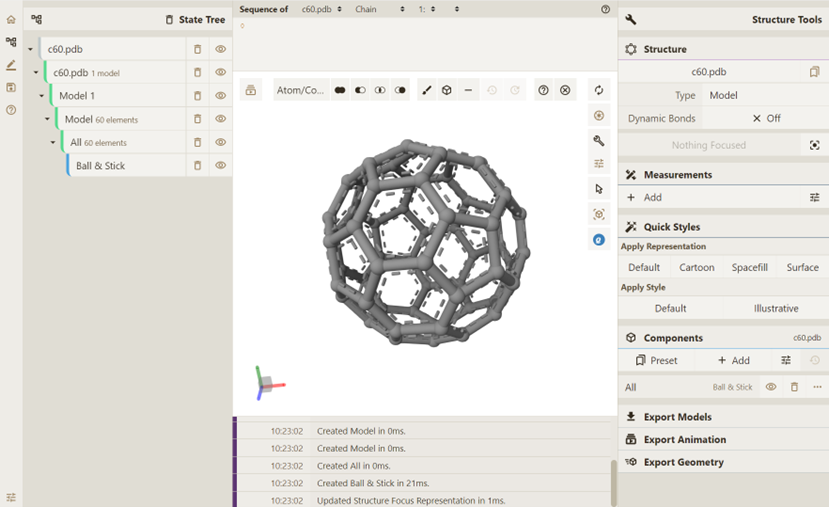
效果如下:
1.3. 分子可视化:在线下载PDB
Qbics-MolStar可以从多种途径下载分子坐标并渲染可视化。以下以PDB为例。
在Qbics-MolStar中键入PDB Id,如
6AP4:
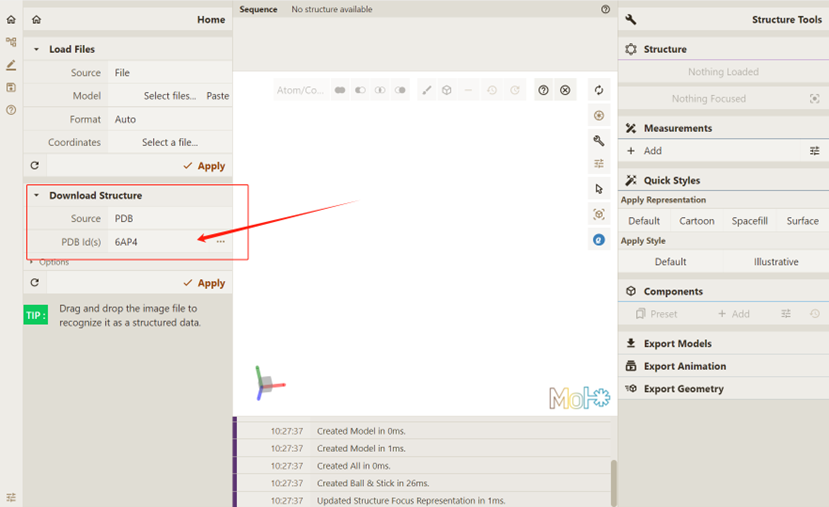
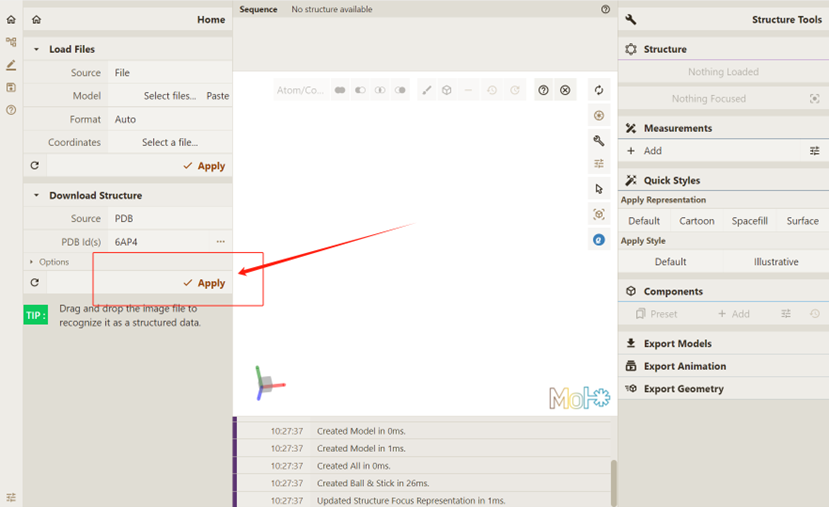
点击 Apply,要求Qbics-MolStar实现可视化:
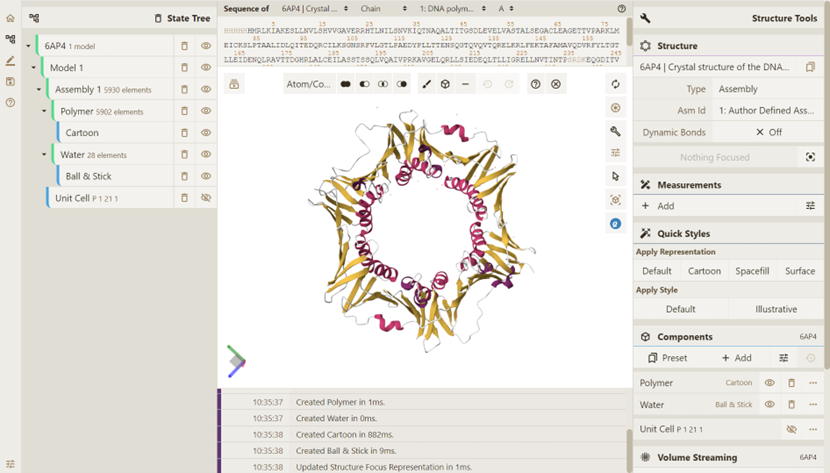
效果如下:
1.4. 分子可视化:SMILES代码
Qbics-MolStar还可以根据SMILES, PubChem等导入数据。我们选择SMILES代码作为另一可视化示例。
修改Source为SMILES而非默认PDB:
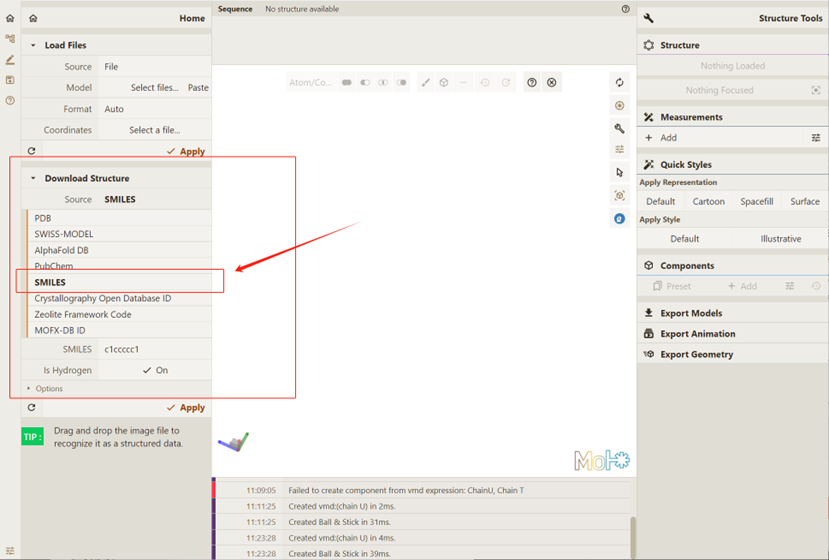
作为本次尝试的SMILES示例,键入要求填写SMILES的位置:
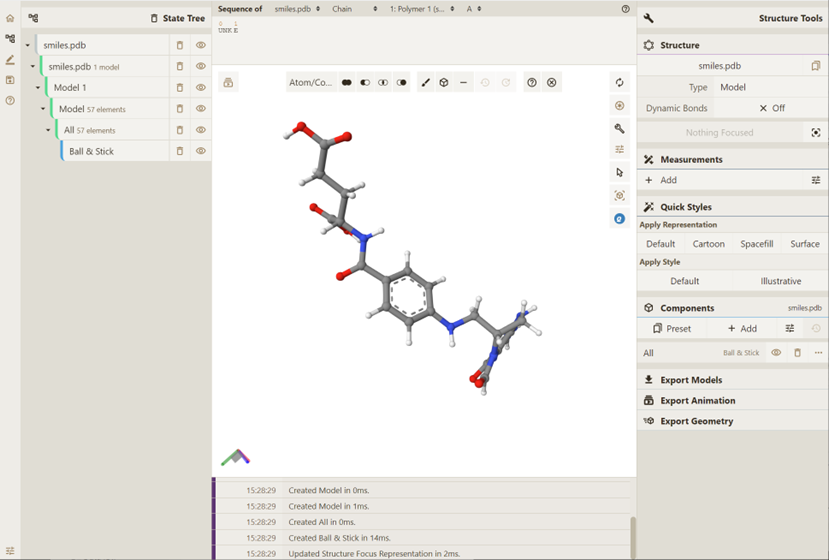
[H]C(=O)N1C(CNC2=CC=C(C=C2)C(=O)N[C@@H](CCC(O)=O)C(O)=O)CNC2=C1C(=O)NC(N)=N2:
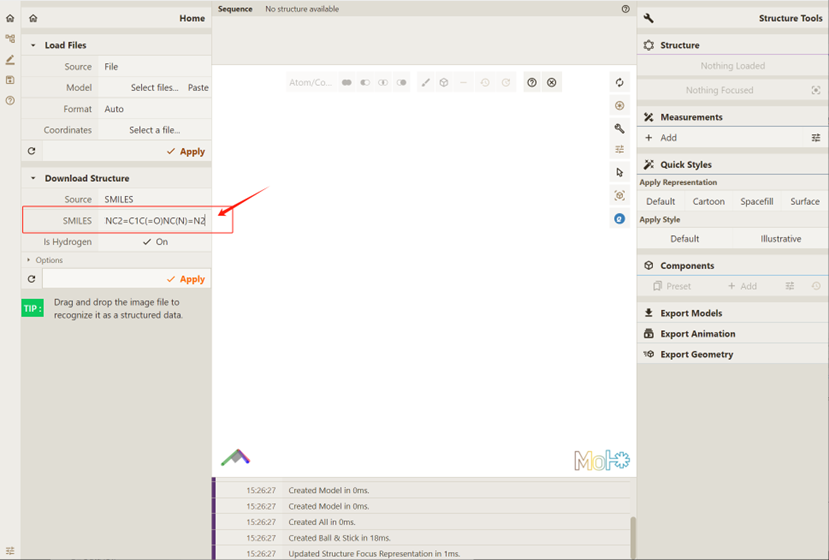
点击 Apply,要求Qbics-MolStar实现可视化:
效果如下: