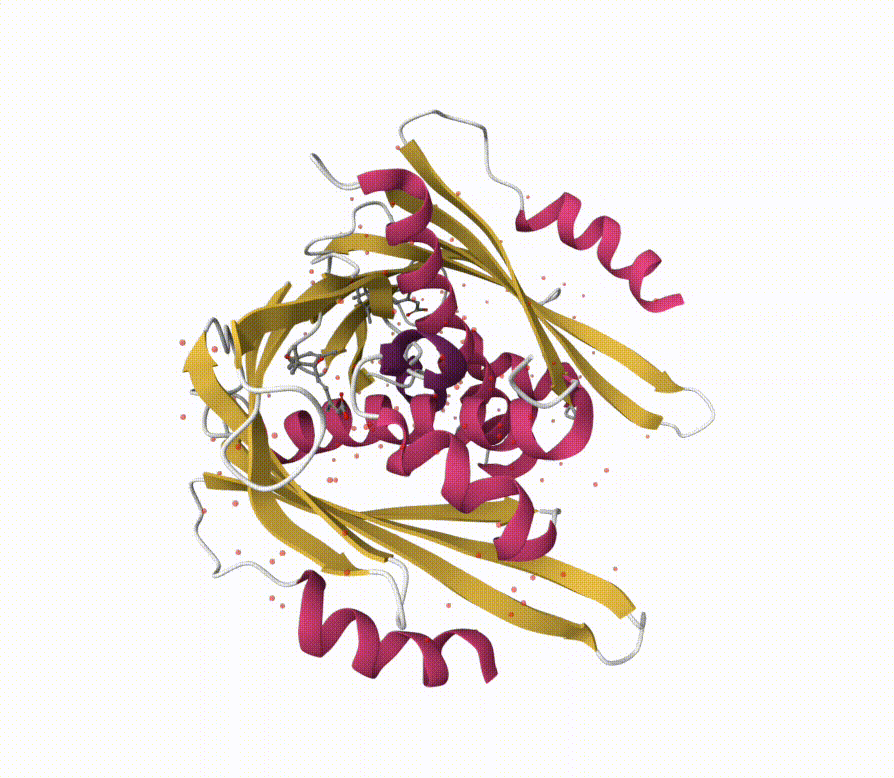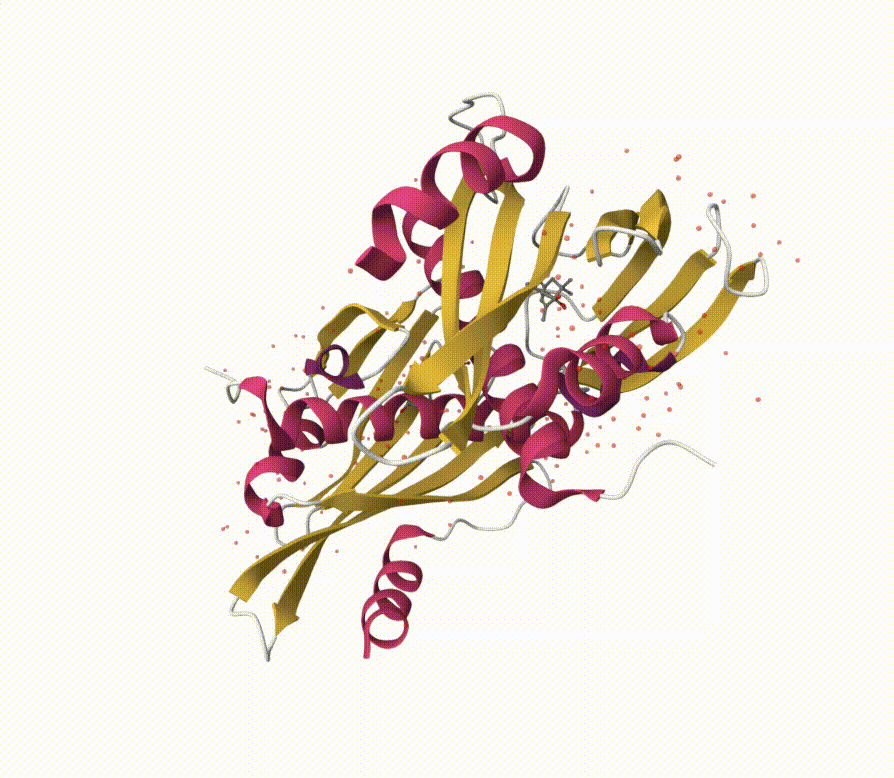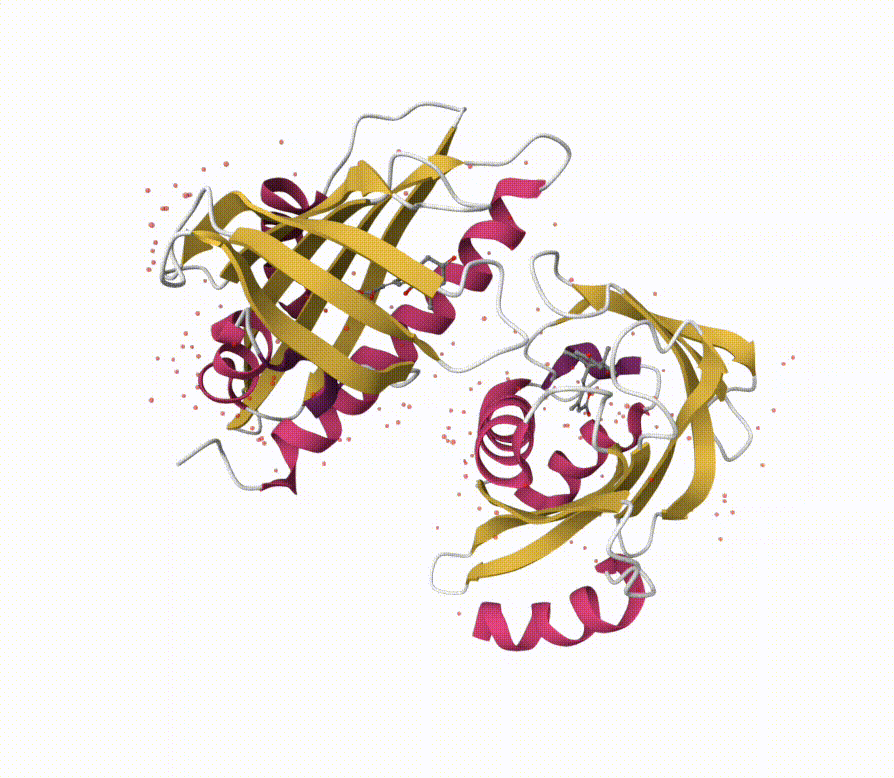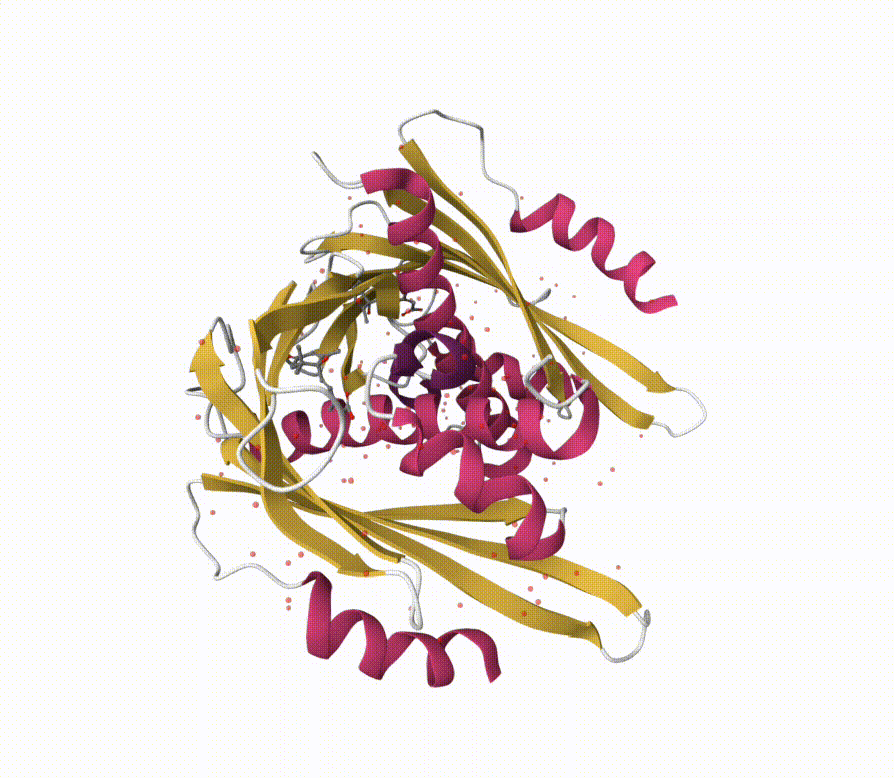2. 可视化轨迹并导出动画
2.1. 以XYZ轨迹文件为例查看轨迹并导出动画
本例中使用的文件可以在这里下载:Files.
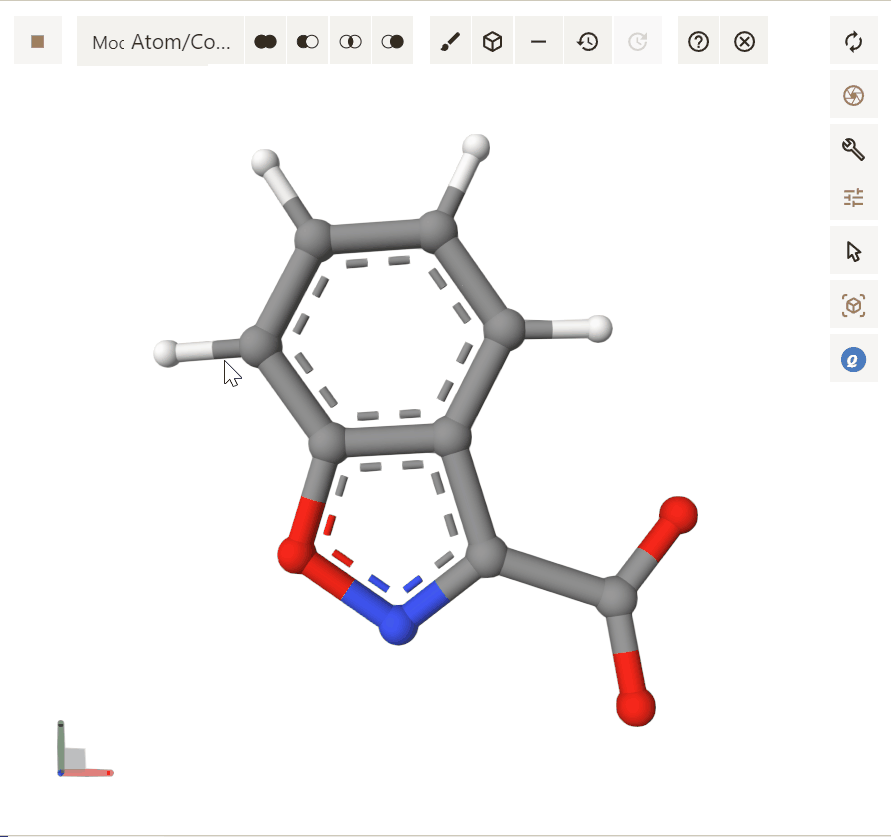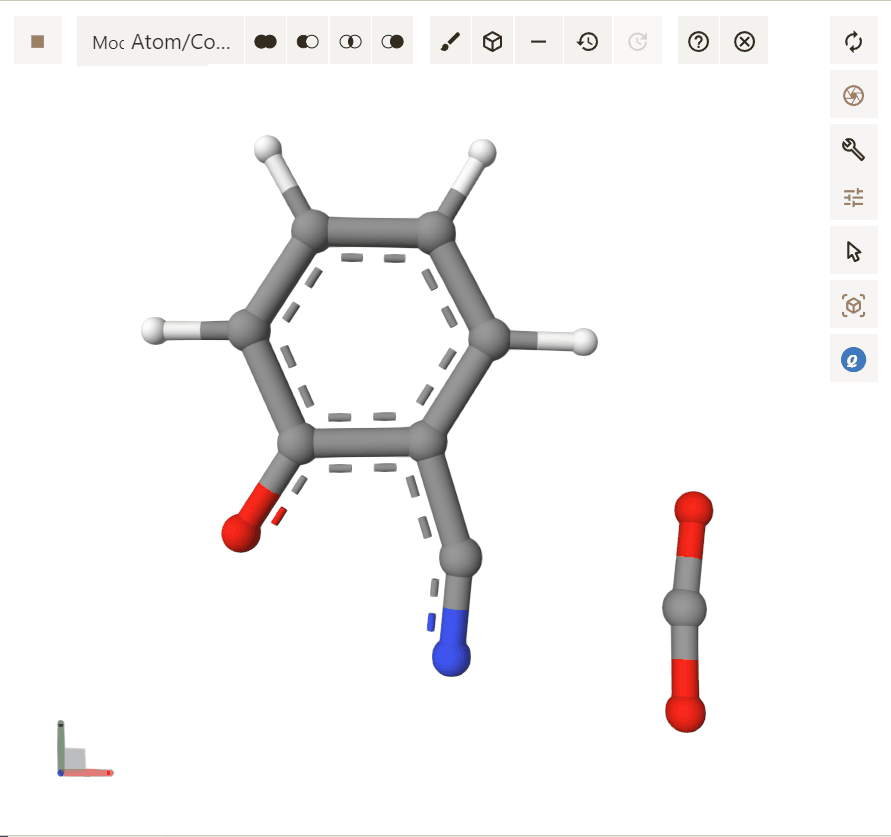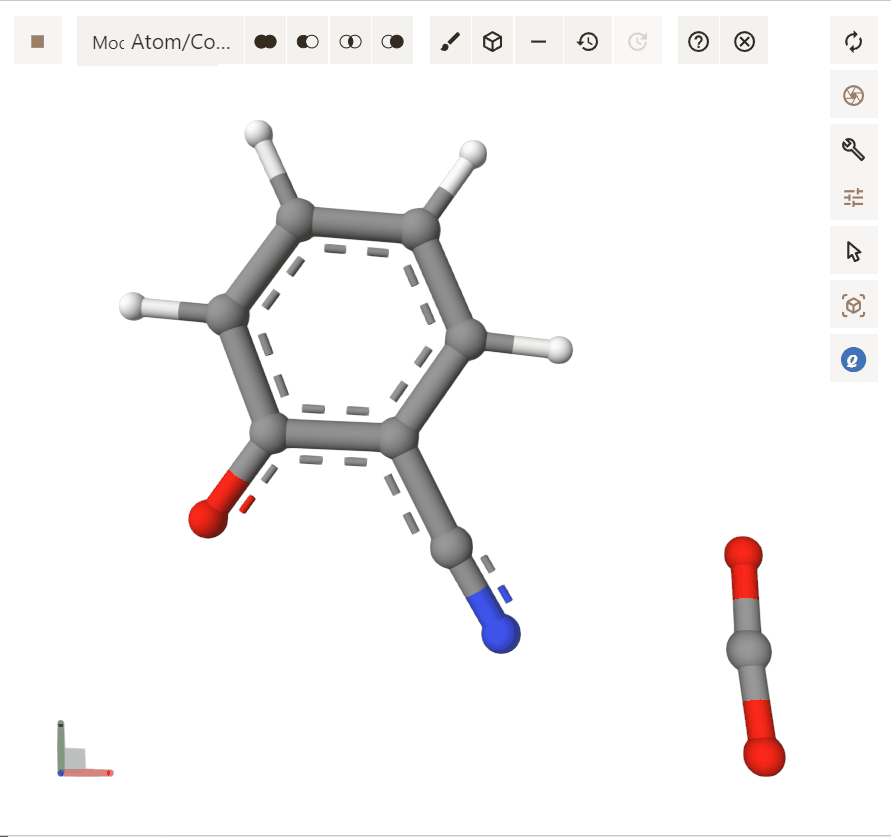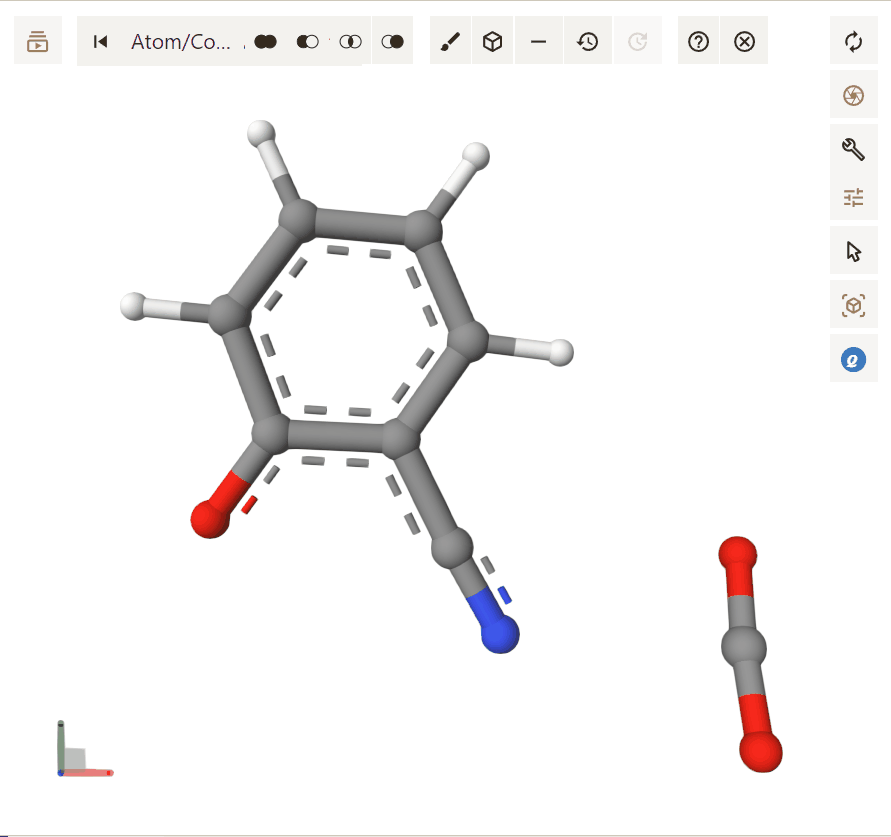
通过访问与安装,可视化文件或SMILES代码的两种方法(拖拽 / 选择文件)使 .xyz 文件在 Qbics-MolStar 界面显示渲染。
请注意,可查看轨迹的前提是当前您所选取的 .xyz 文件存在多帧信息。
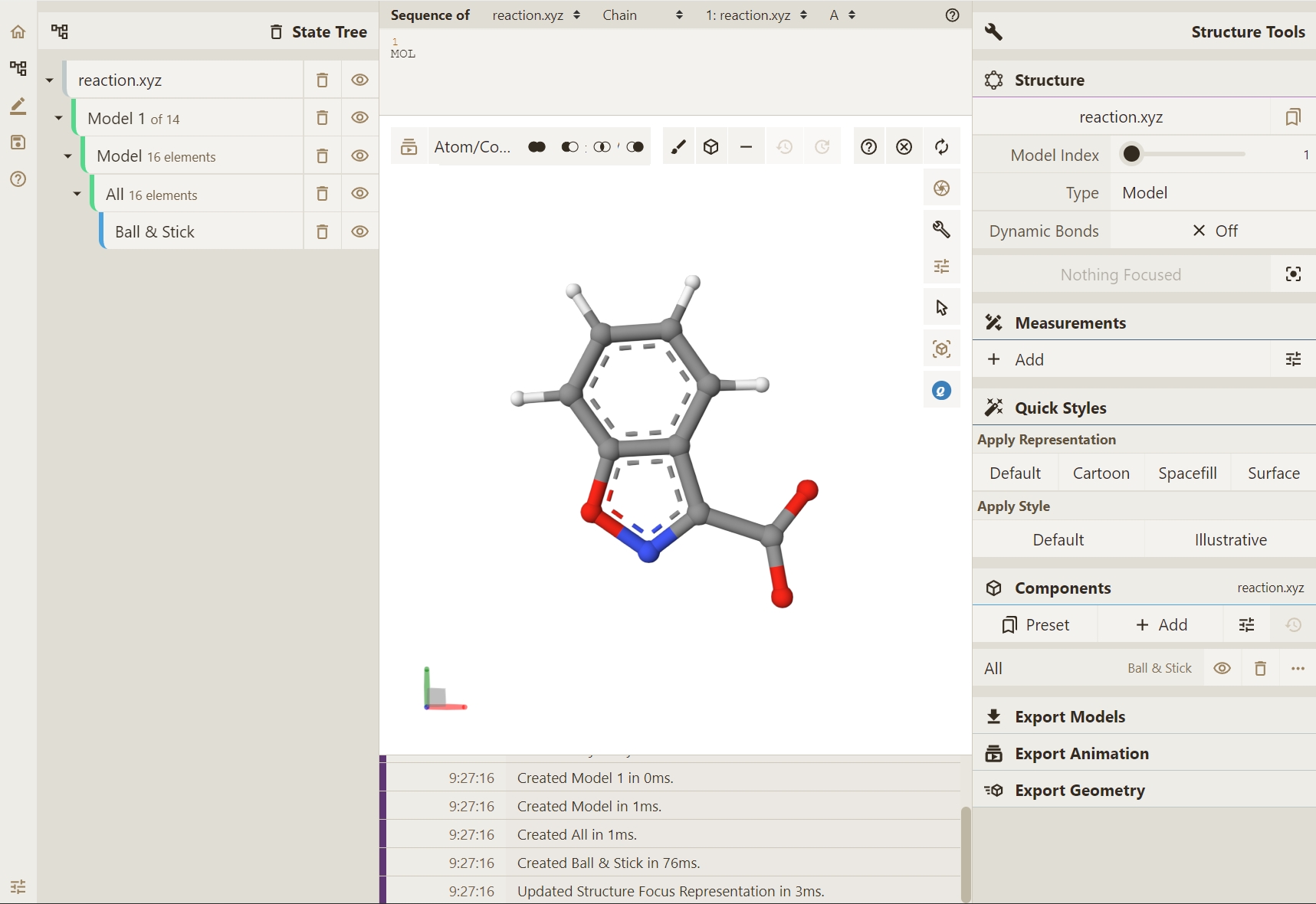
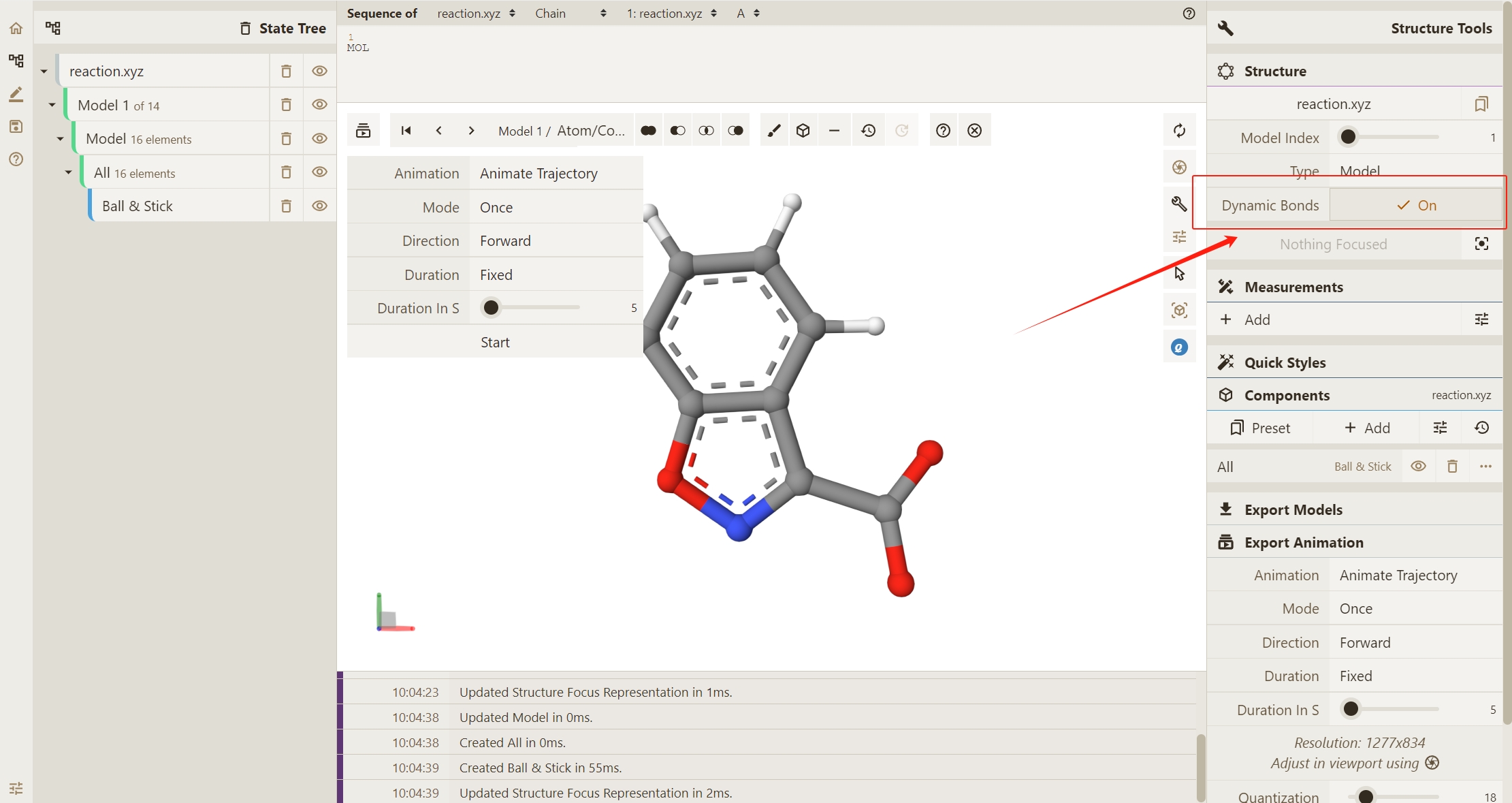
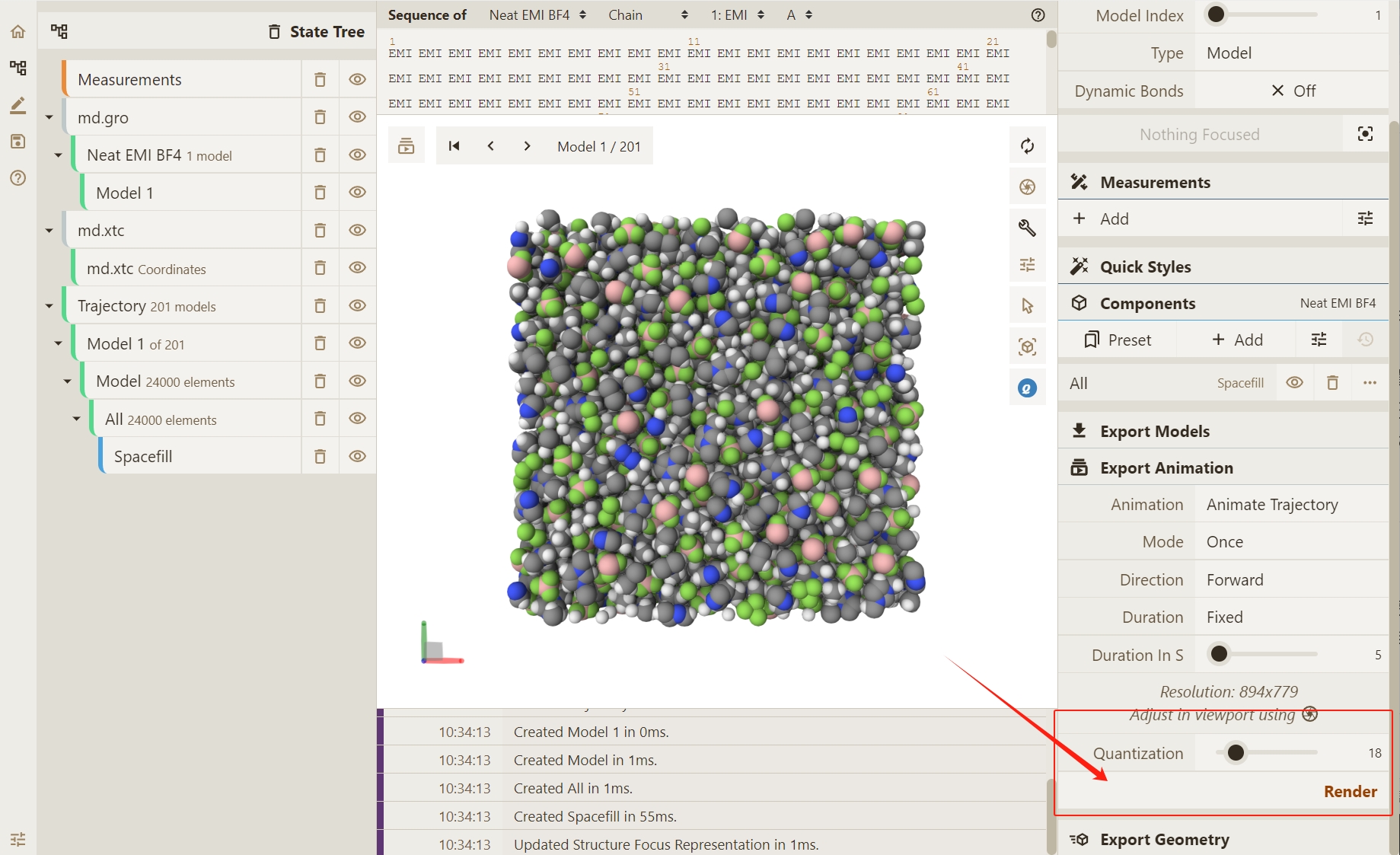
此处,我们开启Dynamic Bonds按钮,以动态观察键连的变化。
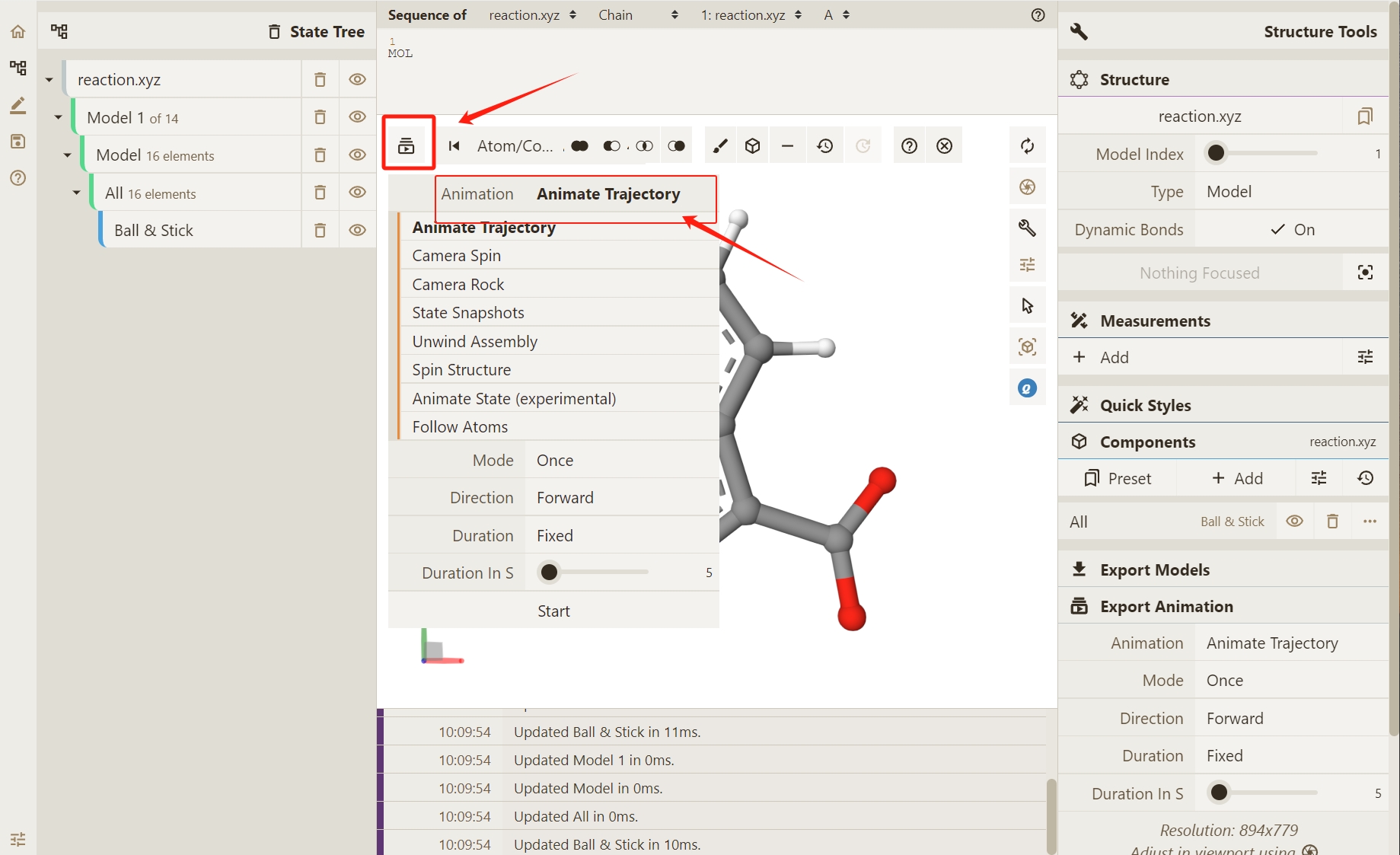
点击图形界面左上角Select Animation按钮,选择不同的轨迹或场景动画演示方式。在此处,我们选择系统默认的 Animate Trajectory 方法(分子模拟轨迹)
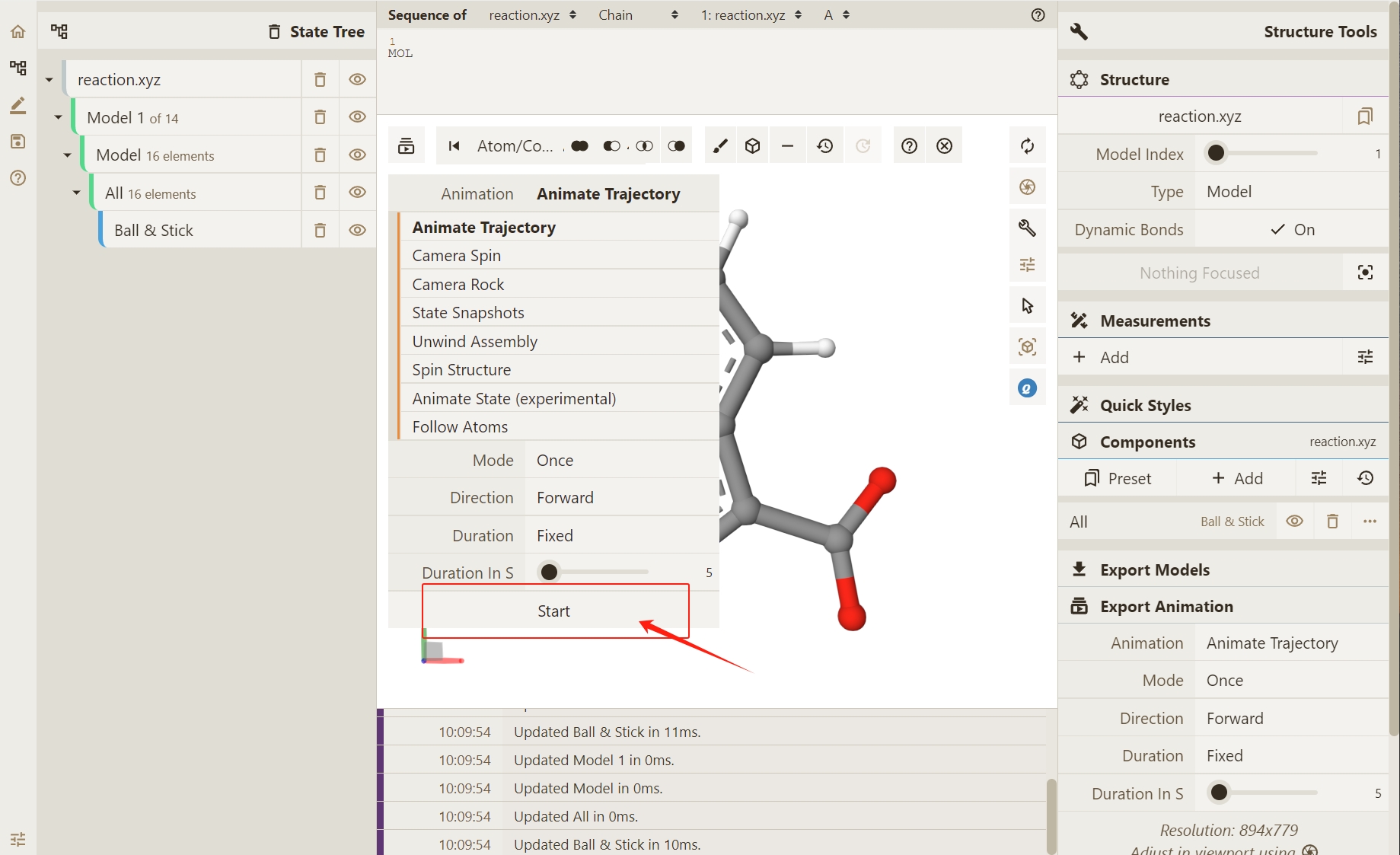
点击Start按钮,渲染轨迹动画。
动画播放效果如下:
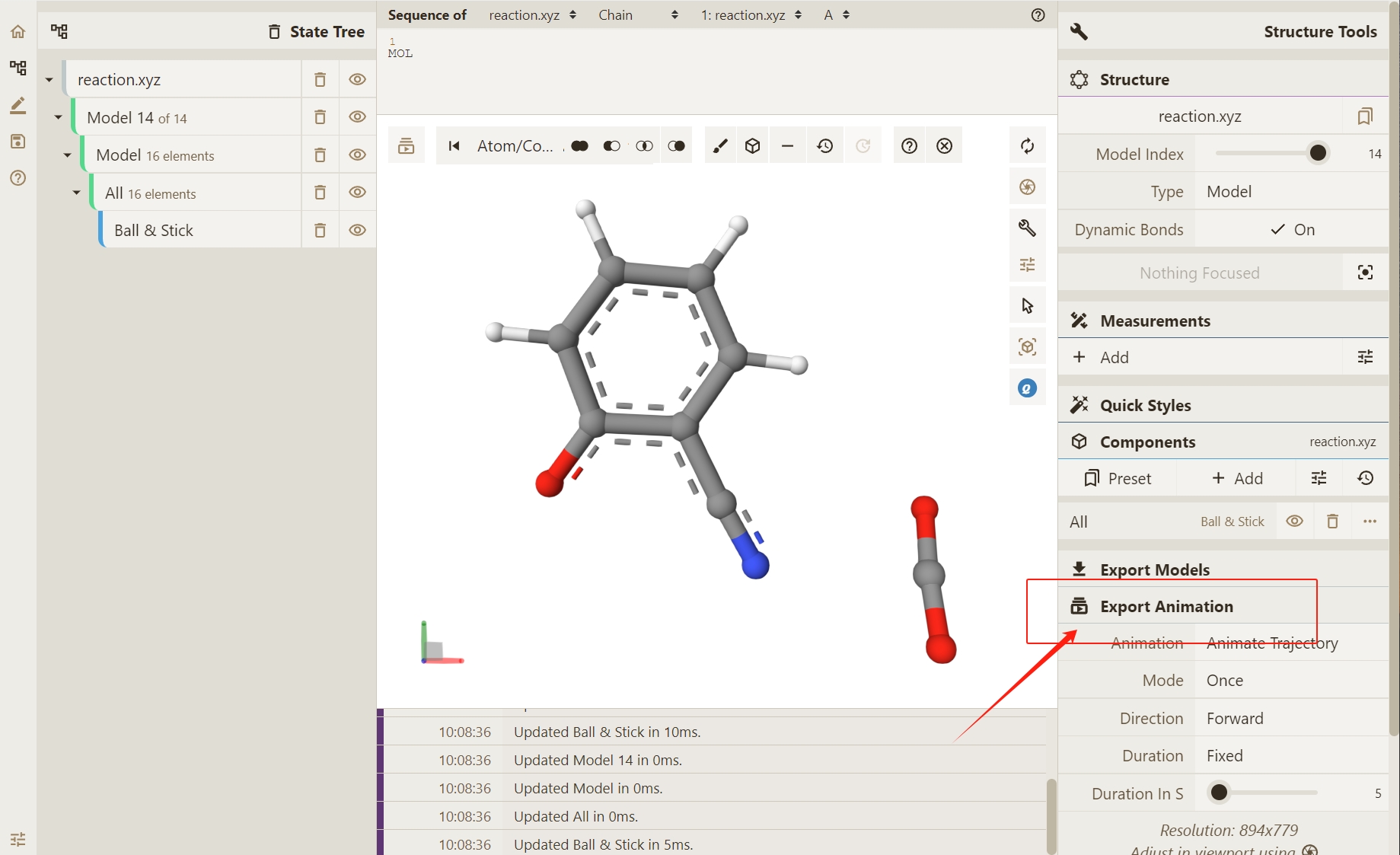
点击Qbics-MolStar界面右下角Export Animation按钮实现轨迹导出功能(此处与Select Animation按钮下的选择情况一致)。
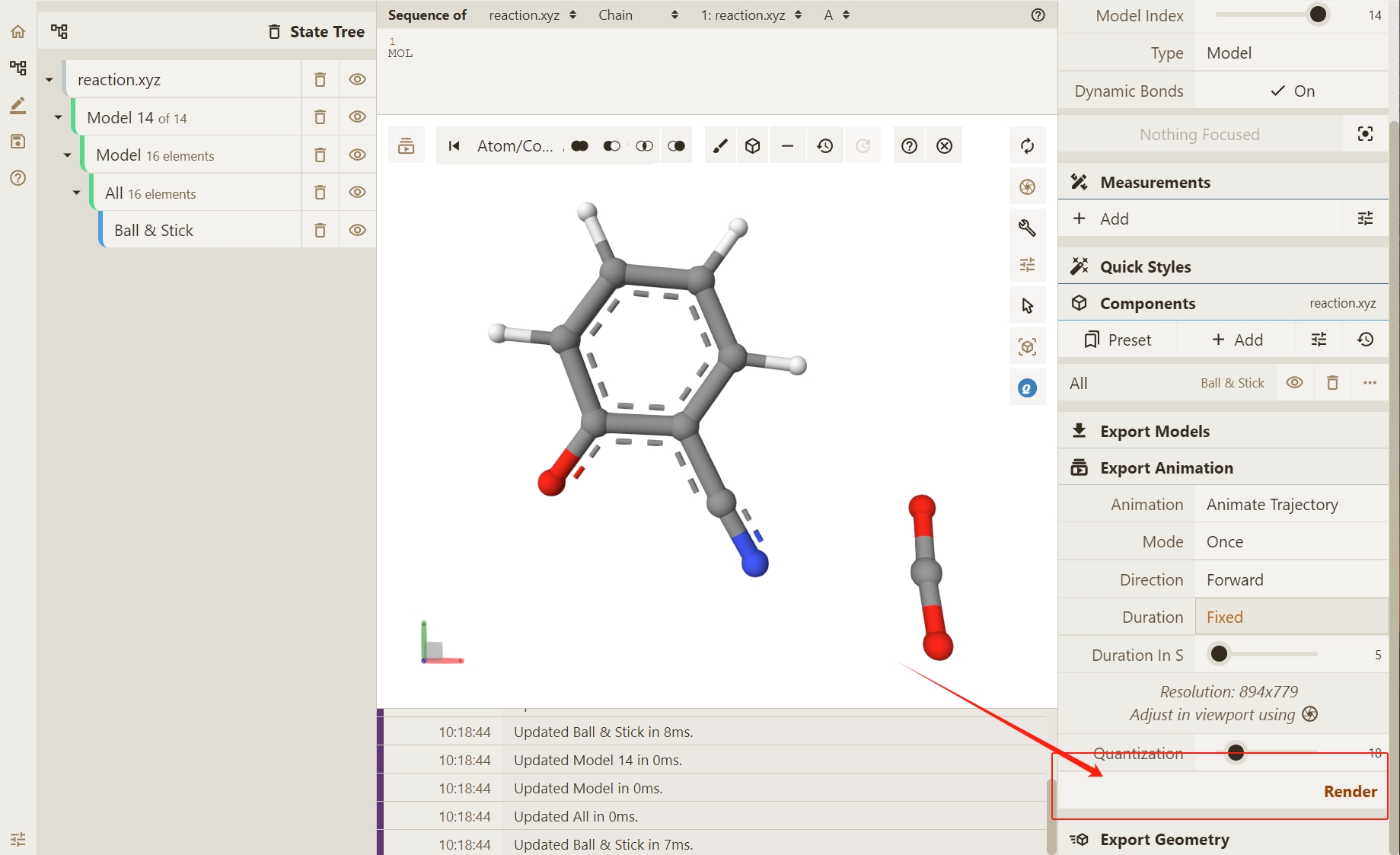
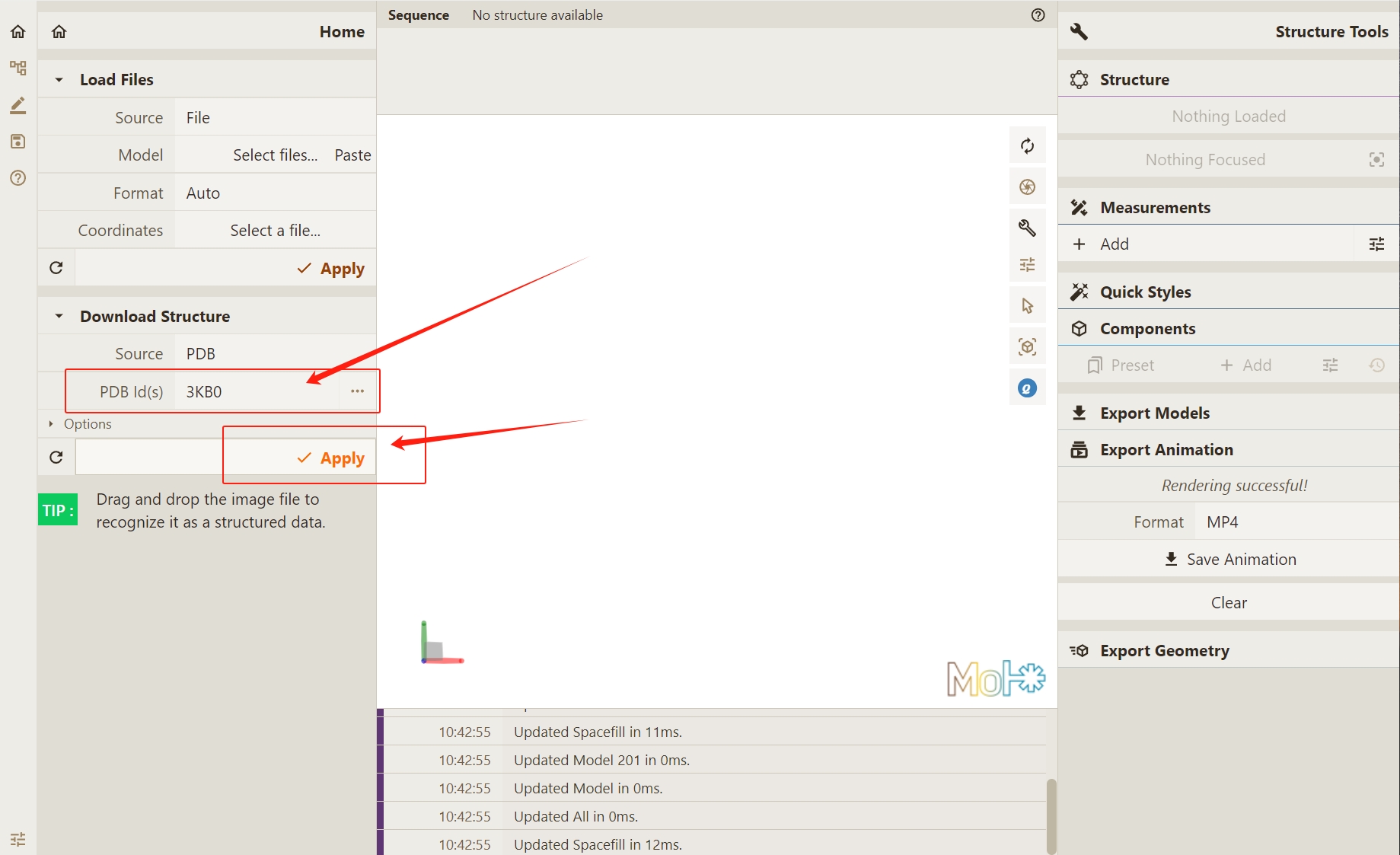
点击Render按钮,使Qbics-Molstar进行轨迹处理。(若提示 Rendering successful 后,您希望更换当前的处理结果,可以点击下方的Clear做清除处理。)
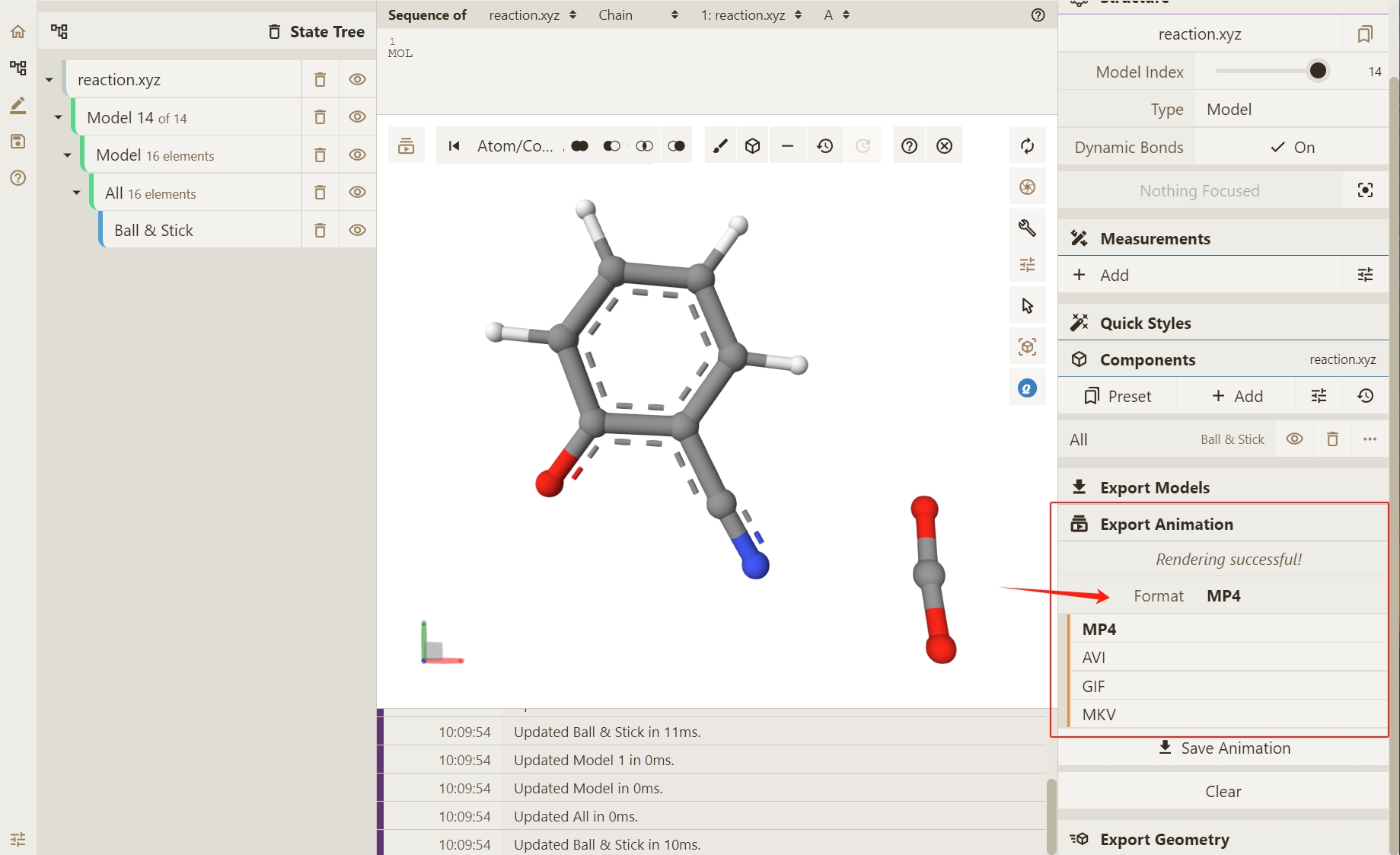
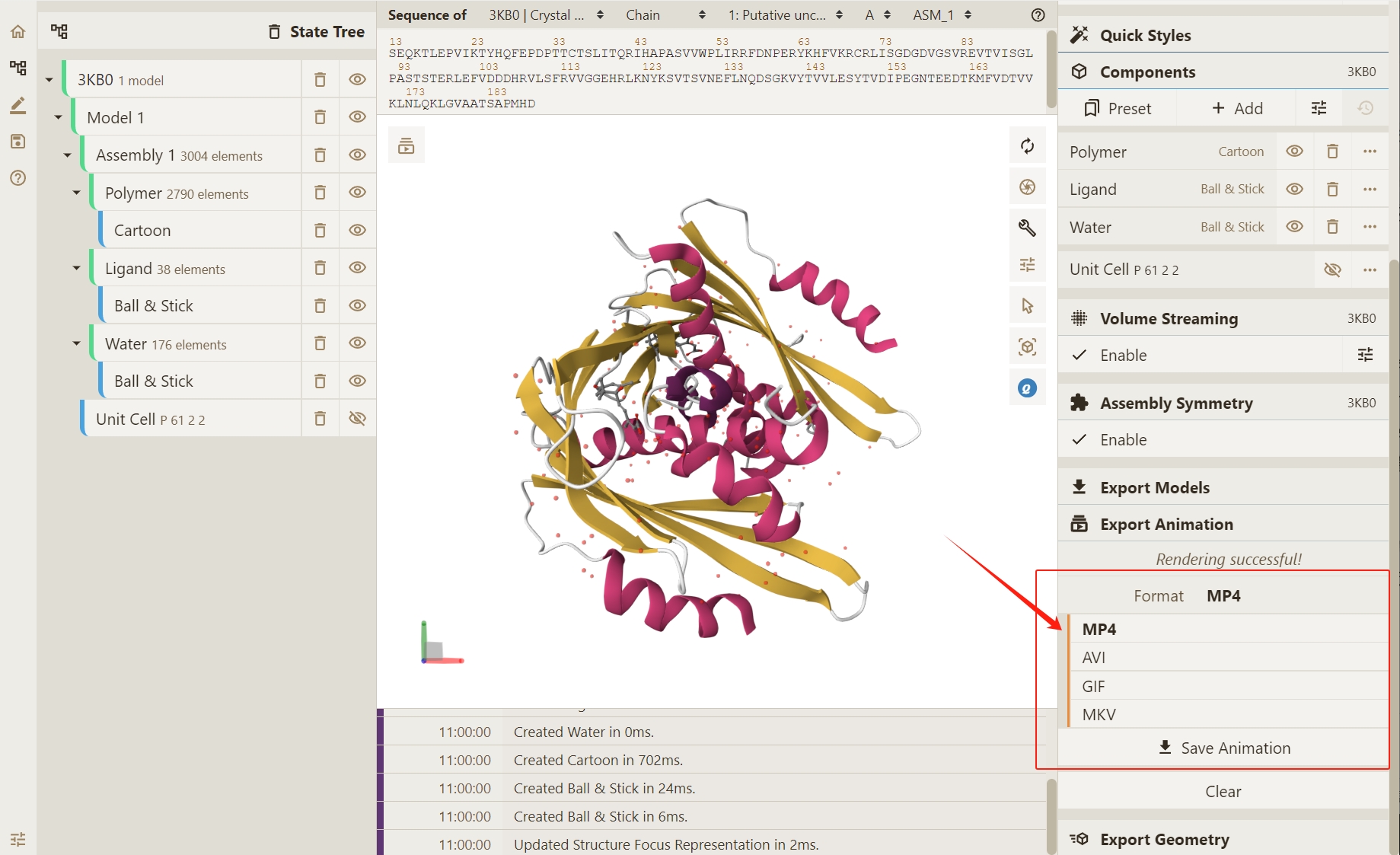
存在 4 种文件类型可以导出:MP4,AVI,GIF,MKV。
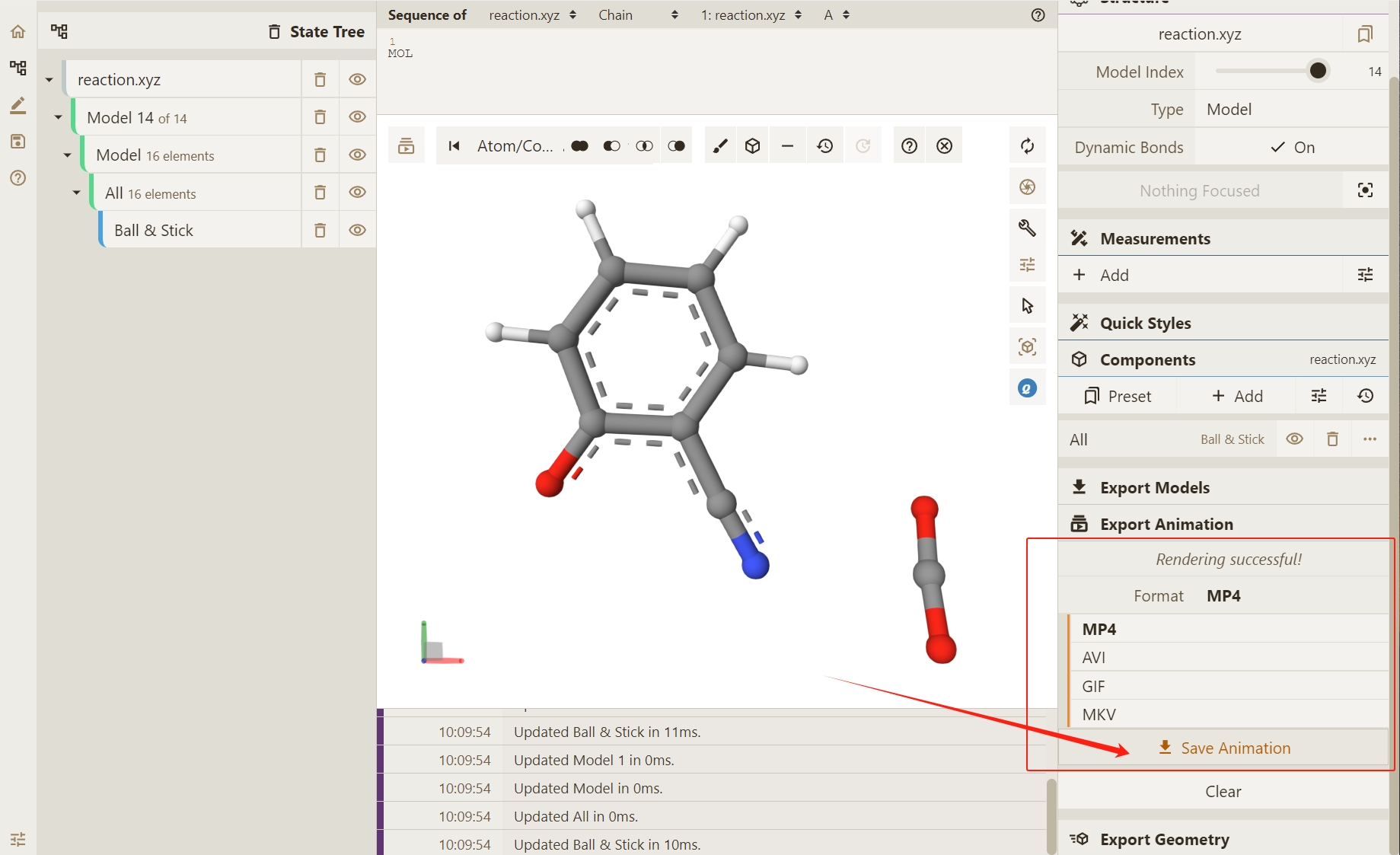
确定希望导出的文件类型后,点击Save Animation导出结果。
此处作为示例,为您展示 GIF 的输出结果。
2.2. 以GRO + XTC文件为例查看轨迹并导出动画
本例中使用的文件可以在这里下载:Files.
XYZ格式中的轨迹既有坐标也有拓扑信息,但是很多分子动力学使用一些二进制格式的轨迹文件,如XTC(GROMACS 压缩格式),它们中并不包含拓扑信息。在 Qbics-MolStar 中,您可以组合任意包含拓扑信息和坐标信息的文件来读轨迹。这里以GRO+XTC来掩饰。
在 Load Files - Model部分选择 .gro 文件作为体系的初始结构信息(选择文件的操作步骤详见访问与安装,可视化文件或SMILES代码):
在 Load Files - Coordinates部分选择 .xtc 文件获取模拟过程中的轨迹坐标信息(选择文件的操作步骤详见访问与安装,可视化文件或SMILES代码):
点击 Apply去命令 Qbics-MolStar 渲染轨迹
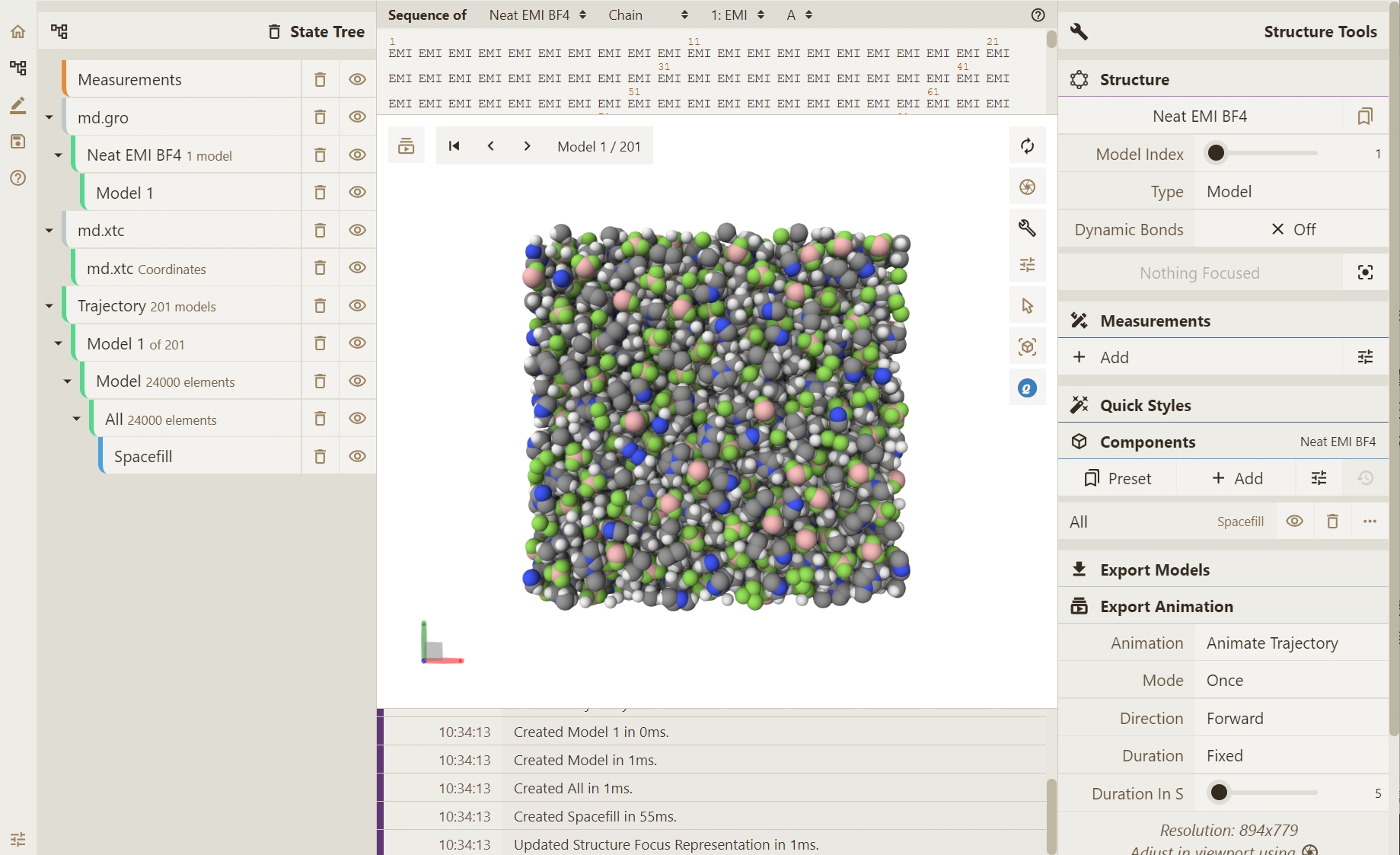
渲染结果如下:
重复本篇教程的上一示例中,关于导出动画的操作流程。
此处同样,以 GIF 文件格式作为示例为您展示轨迹动画结果。

2.3. 以蛋白质为例导出旋转动画
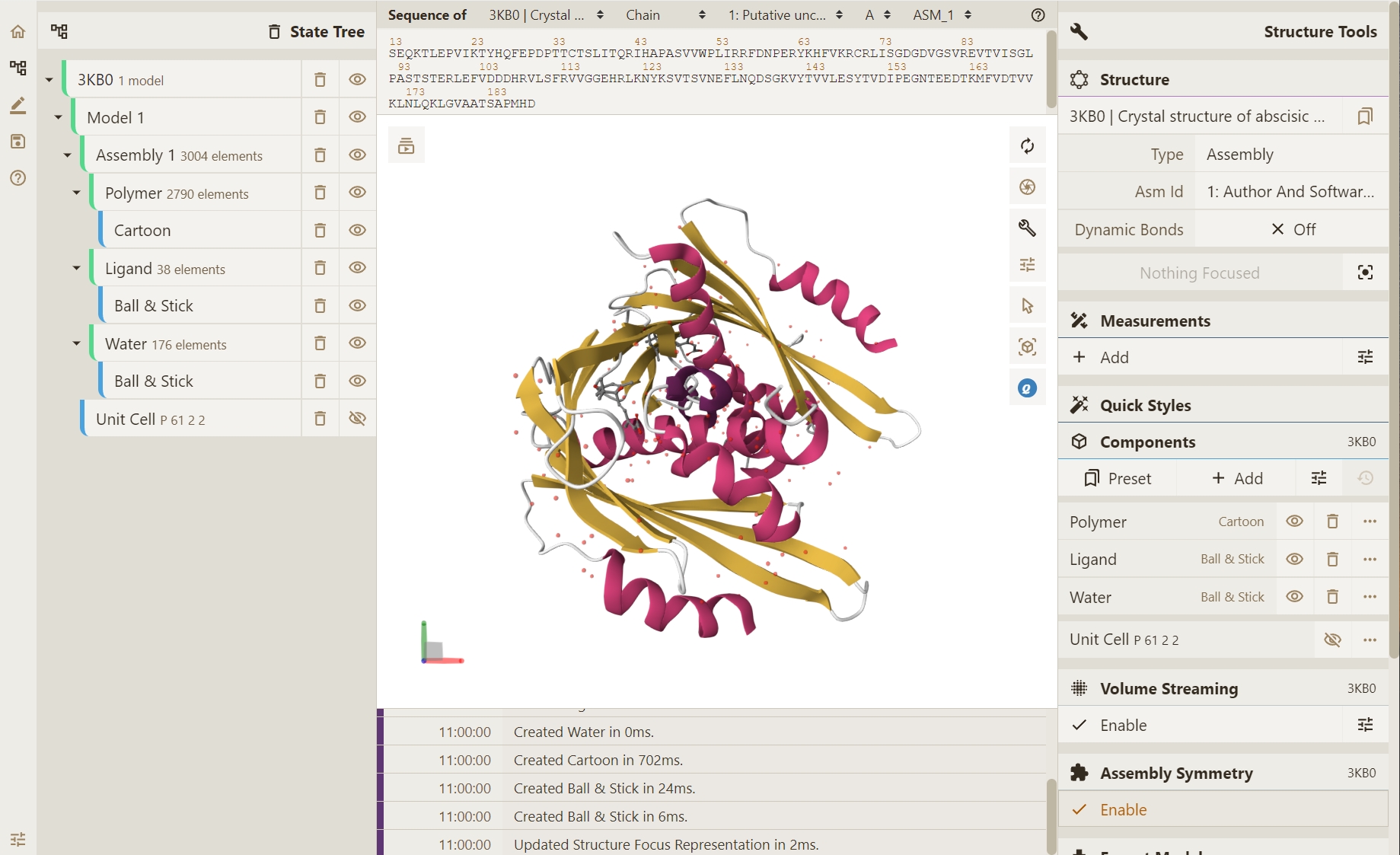
在此处我们以 3KB0 蛋白质为例。
首先,我们在 PDB Id位置键入
3KB0, 并点击 Apply获取蛋白质文件。
渲染结果如下:
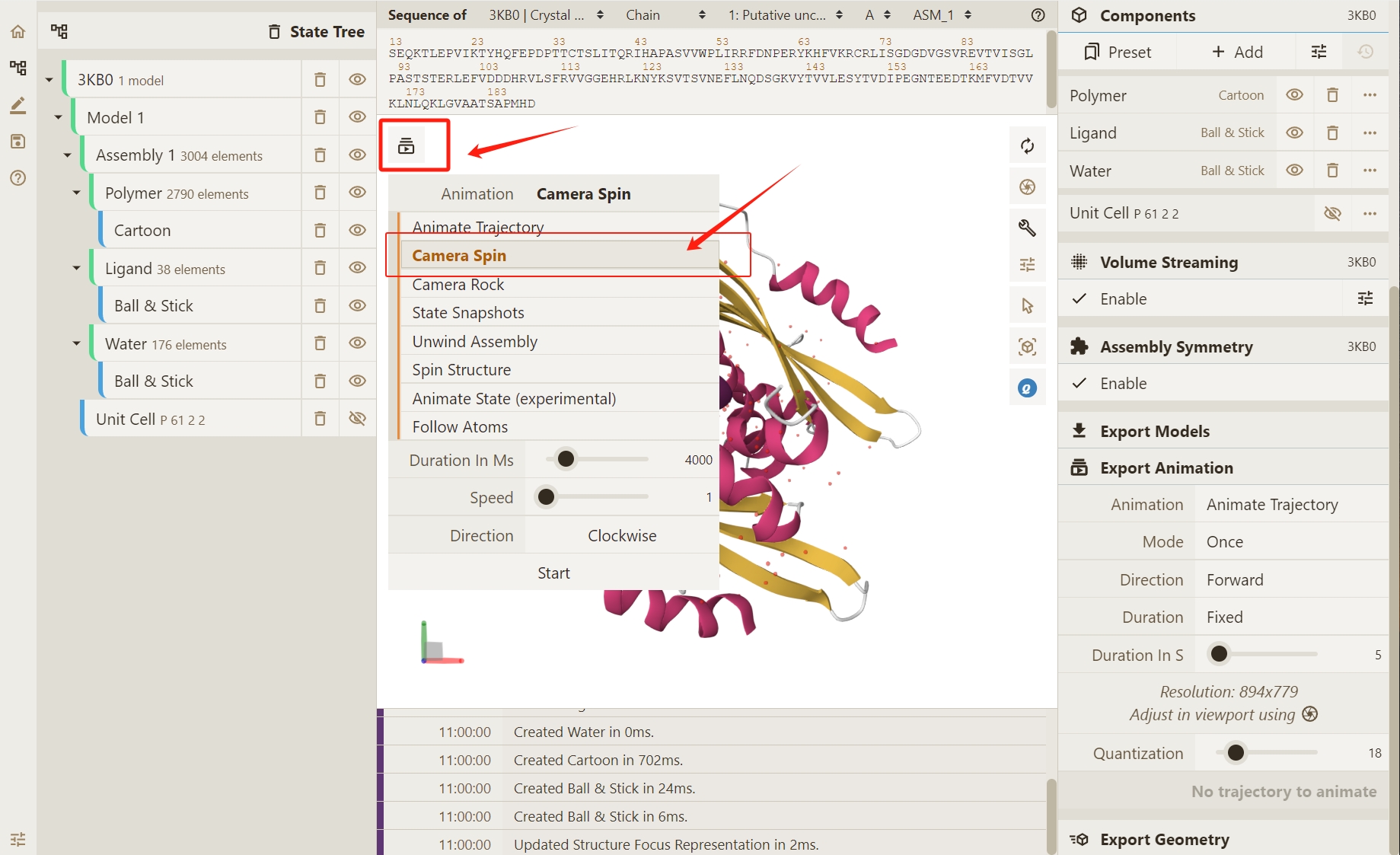
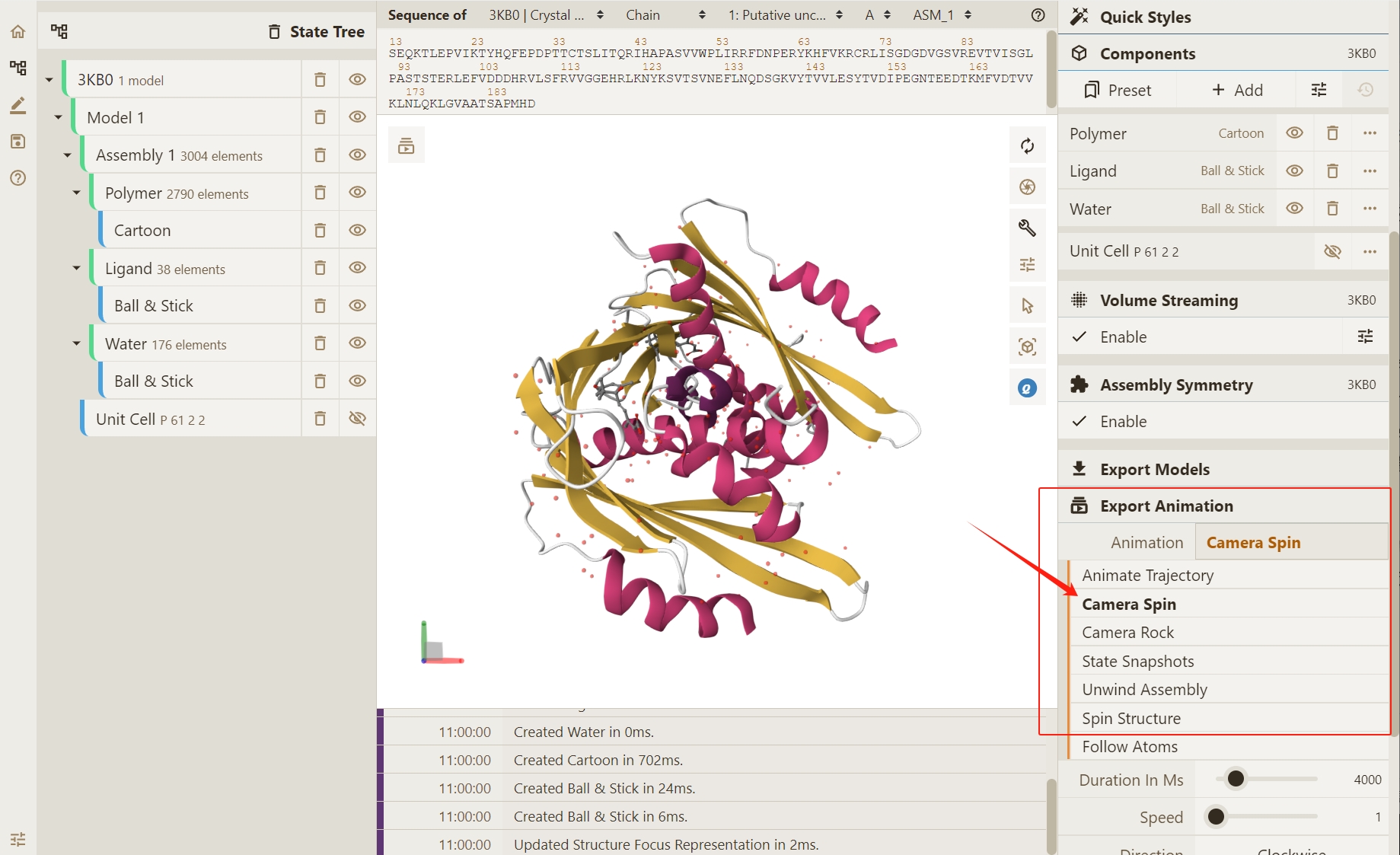
针对蛋白质体系,我们在 Animation选项中选择 :guilabel:`Camera Spin`(旋转相机视角)来进行动画演示
在 Export Animation部分,更改默认 Animate Trajectory 为 Camera Spin。
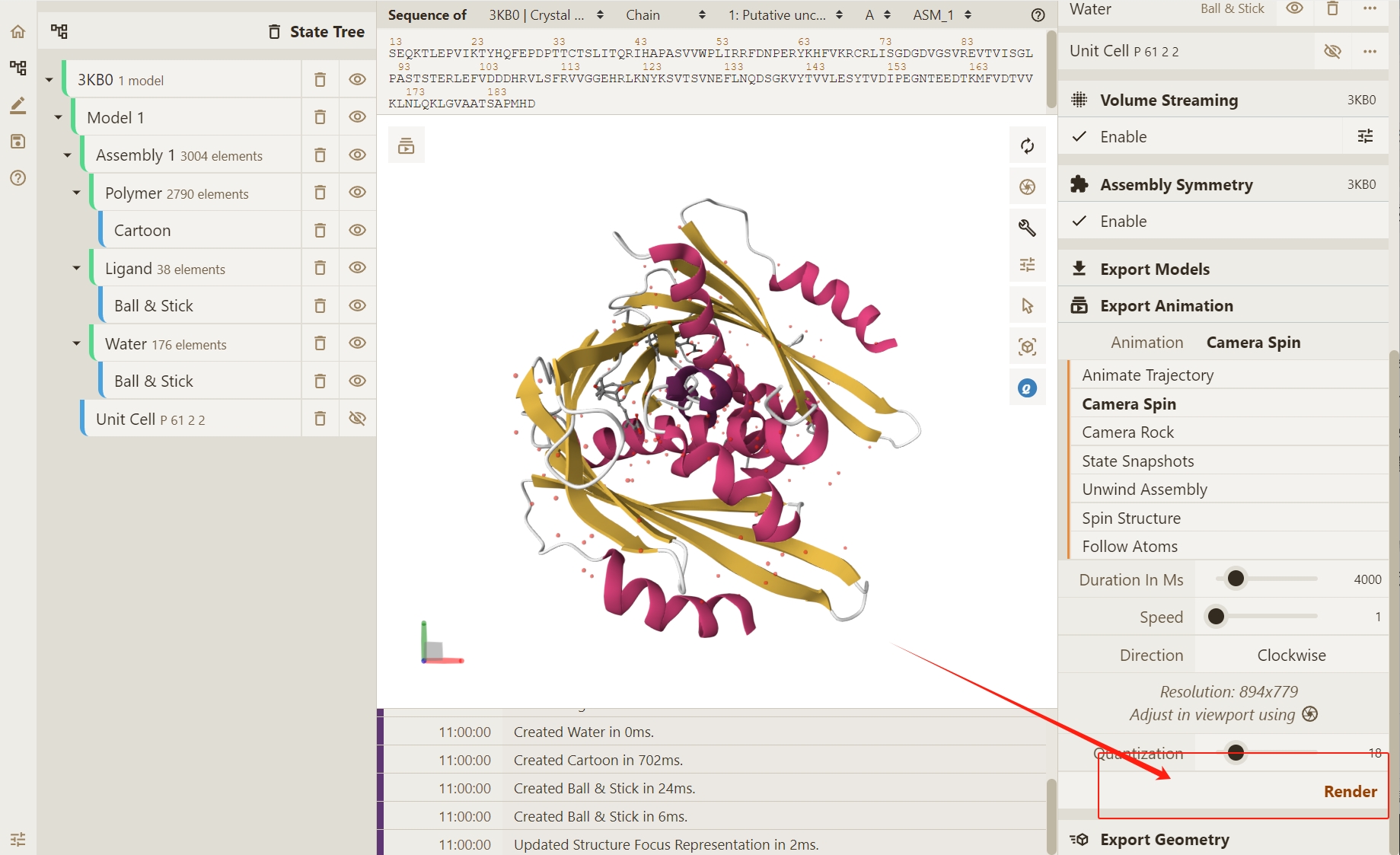
点击 Render按钮,使 Qbics-Molstar 实现轨迹渲染:
我们在此处同样选取 GIF 格式的动画,并将输出文件作为示例进行展示。